Dzień 2 - Bazowa grafika - ciąg dalszy
Bazowa grafika - ciąg dalszy
Funkcja axis
Komentarz: pierwszy parametr axis określa położenie osi: 1- dół, 2- lewa strona, 3 - góra, 4 - prawa strona.
a<-c(1,2,3,4,5,6,7)
b<-c(23,3,2,4,5,2,-2)
plot(a,b,axes=FALSE,type="o",col="blue",ann=FALSE)
axis(4, las=1, at=10*0:range(b)[2])
axis(3, at=1:7, lab=c("Pn","Wt","Śr","Czw","Pt","So","Nd"))
box()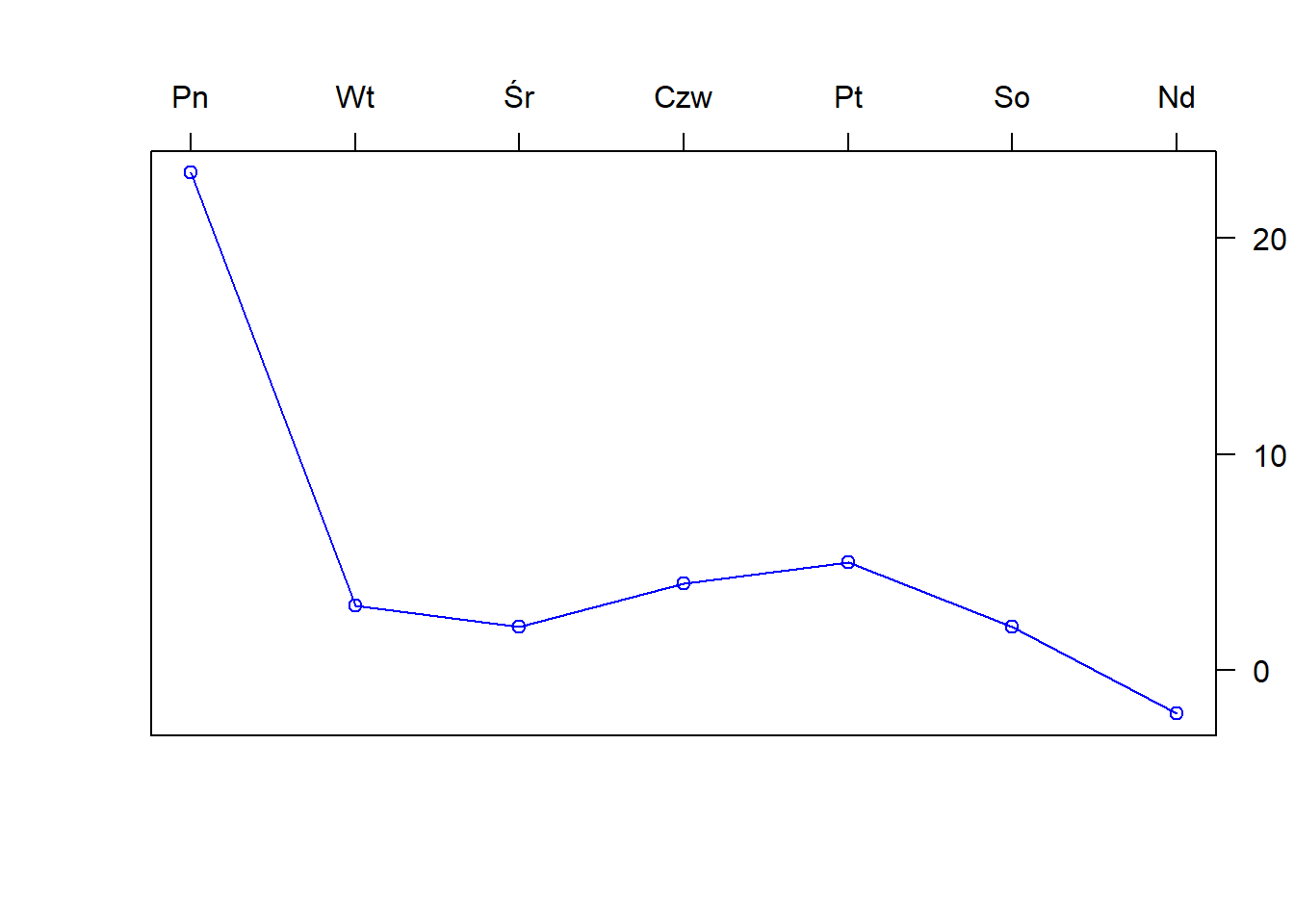
Parametr at - kontrola nad podziałką
Jeśli w parametrze at chcemy mieć kontrolę nad tym co będzie, możemy dodać tam ręcznie konkretny wektor. Ale musimy pamiętać o marginesach.
a<-c(1,2,3,4,5,6,7)
b<-c(23,3,2,4,5,2,-2)
plot(a,b,axes=FALSE,type="o",col="blue",ann=FALSE)
axis(2, las=1, at=c(0,5,17,20,23))
axis(1, at=1:7, lab=c("Pn","Wt","Śr","Czw","Pt","So","Nd"))
box()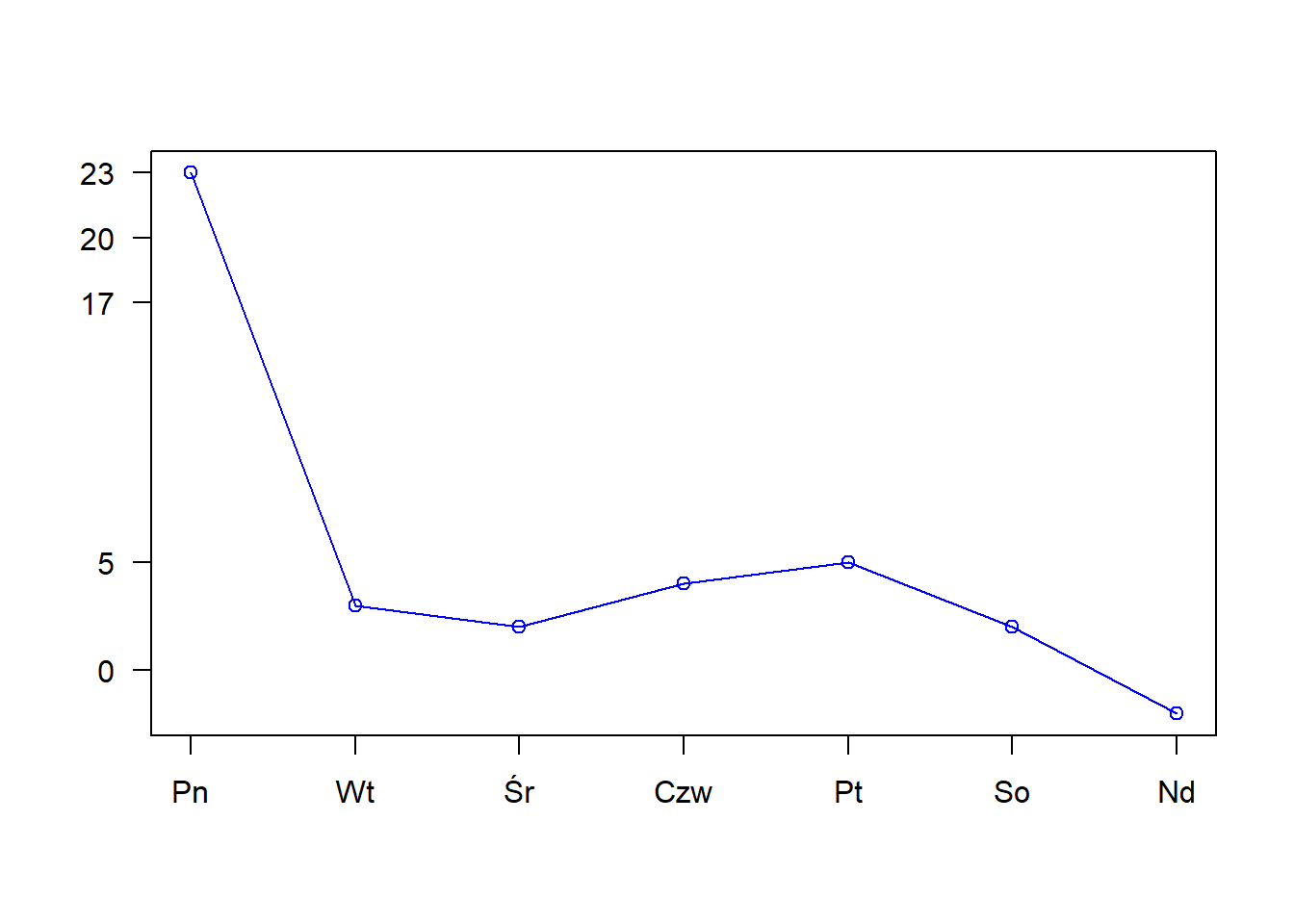
Wykresy dwuosiowe
Wykresy dwuosiowe należy stosować z dużą rozwagą. Najprościej użyć parametru new ustawionego na TRUE. Parametry xaxt i yaxt dotyczą odpowienich podziałek na osiach. Przykładowo:
x<-c(1,2,3,4,5)
y1<-c(5,4,6,7,4)
y2<-c(100,101,103,99,98)
plot(x,y1,type="l",col="red")
par(new=TRUE)
plot(x, y2,,type="l",col="blue",xaxt="n", yaxt="n",xlab="",ylab="")
axis(4)
legend("topleft",col=c("red","blue"),lty=1,legend=c("y1","y2"))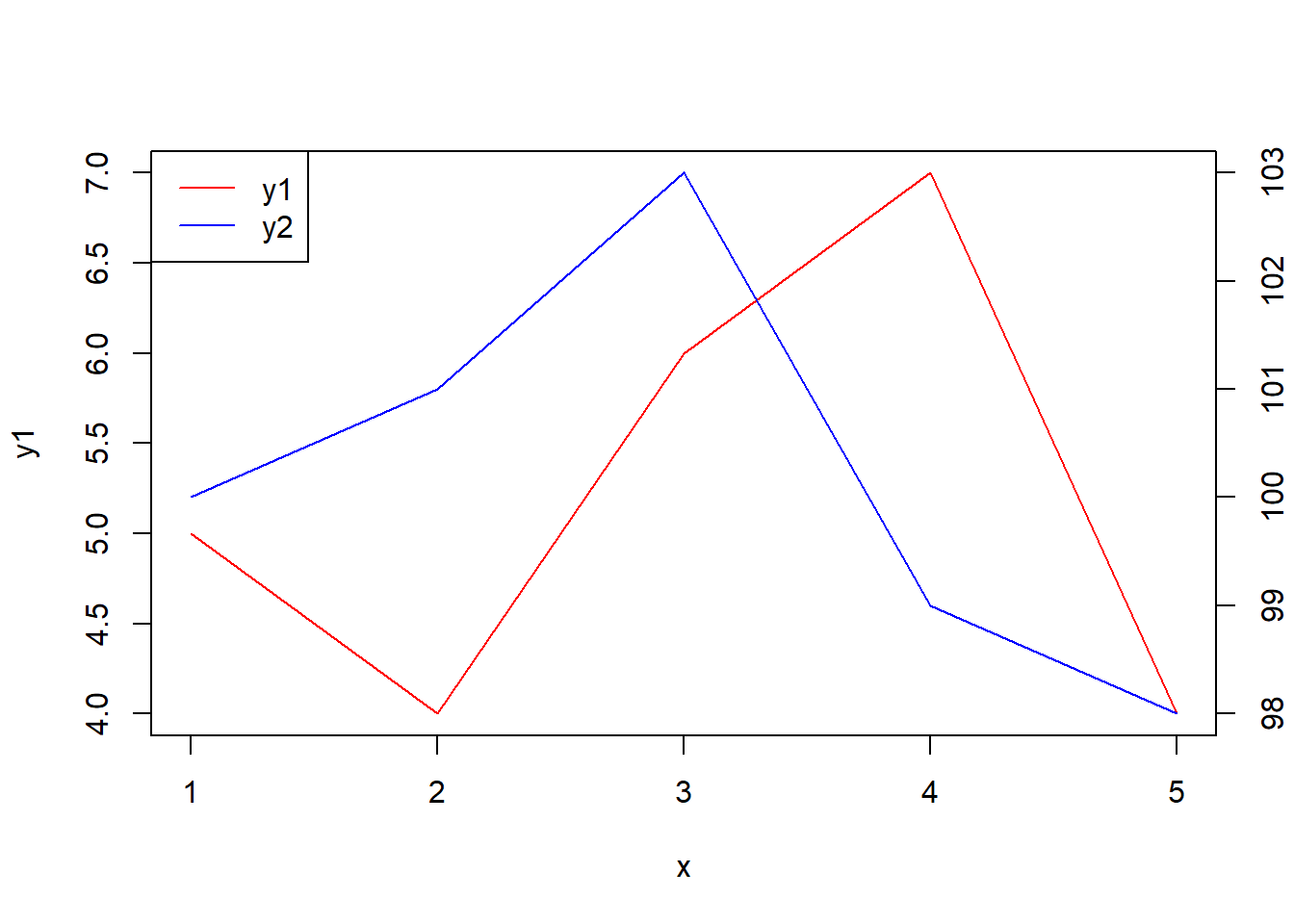
A jak dodać etykietę prawej osi y?
old_par <- par(no.readonly = TRUE)
par(mar=c(5,4,4,5)+.1)
x<-c(1,2,3,4,5)
y1<-c(5,4,6,7,4)
y2<-c(100,101,103,99,98)
plot(x,y1,type="l",col="red")
par(new=TRUE)
plot(x, y2,,type="l",col="blue",xaxt="n", yaxt="n",xlab="",ylab="")
axis(4)
legend("topleft",col=c("red","blue"),lty=1,legend=c("y1","y2"))
mtext("y2",side=4,line=3)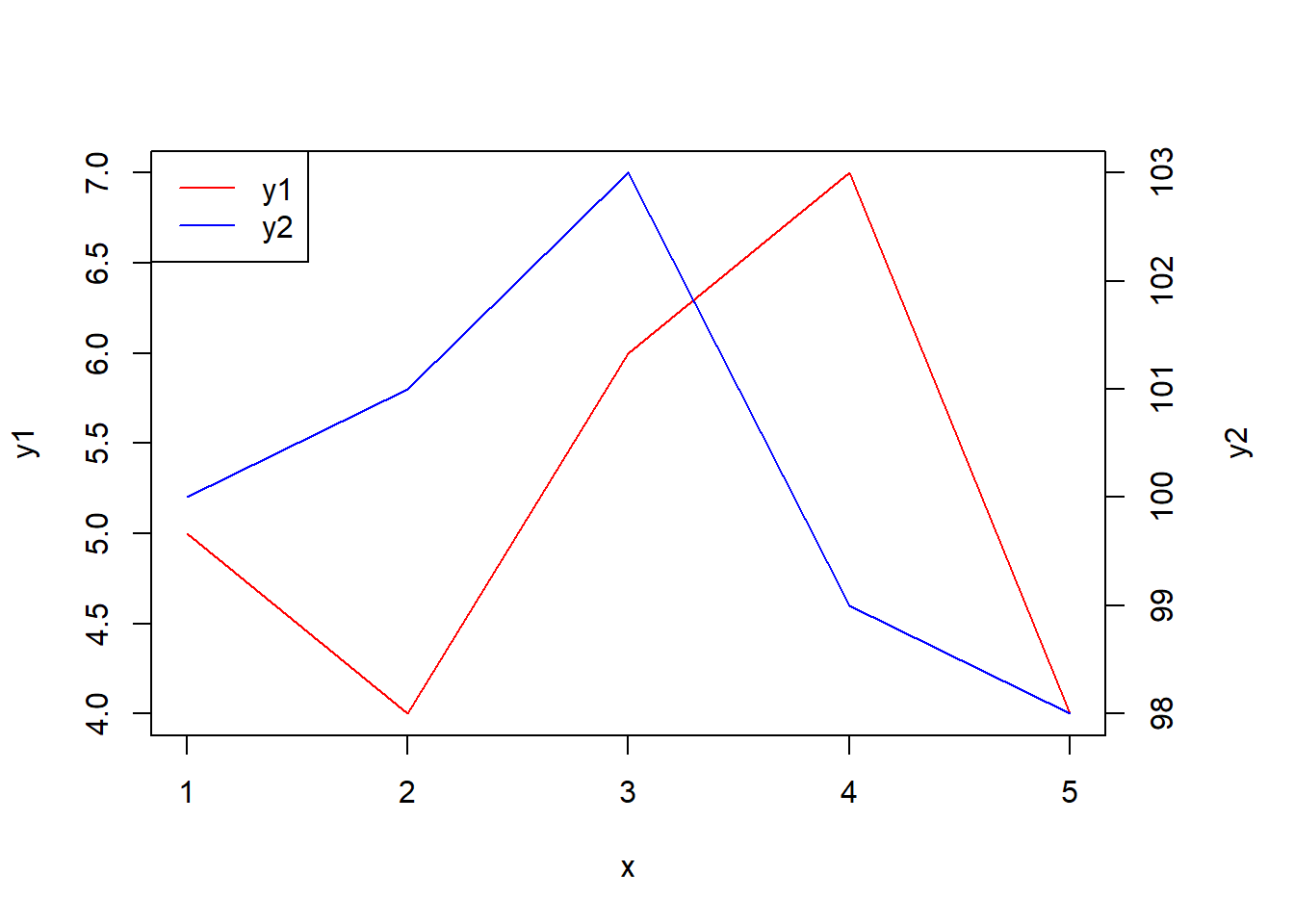
par(old_par)Kolory - jeszcze raz
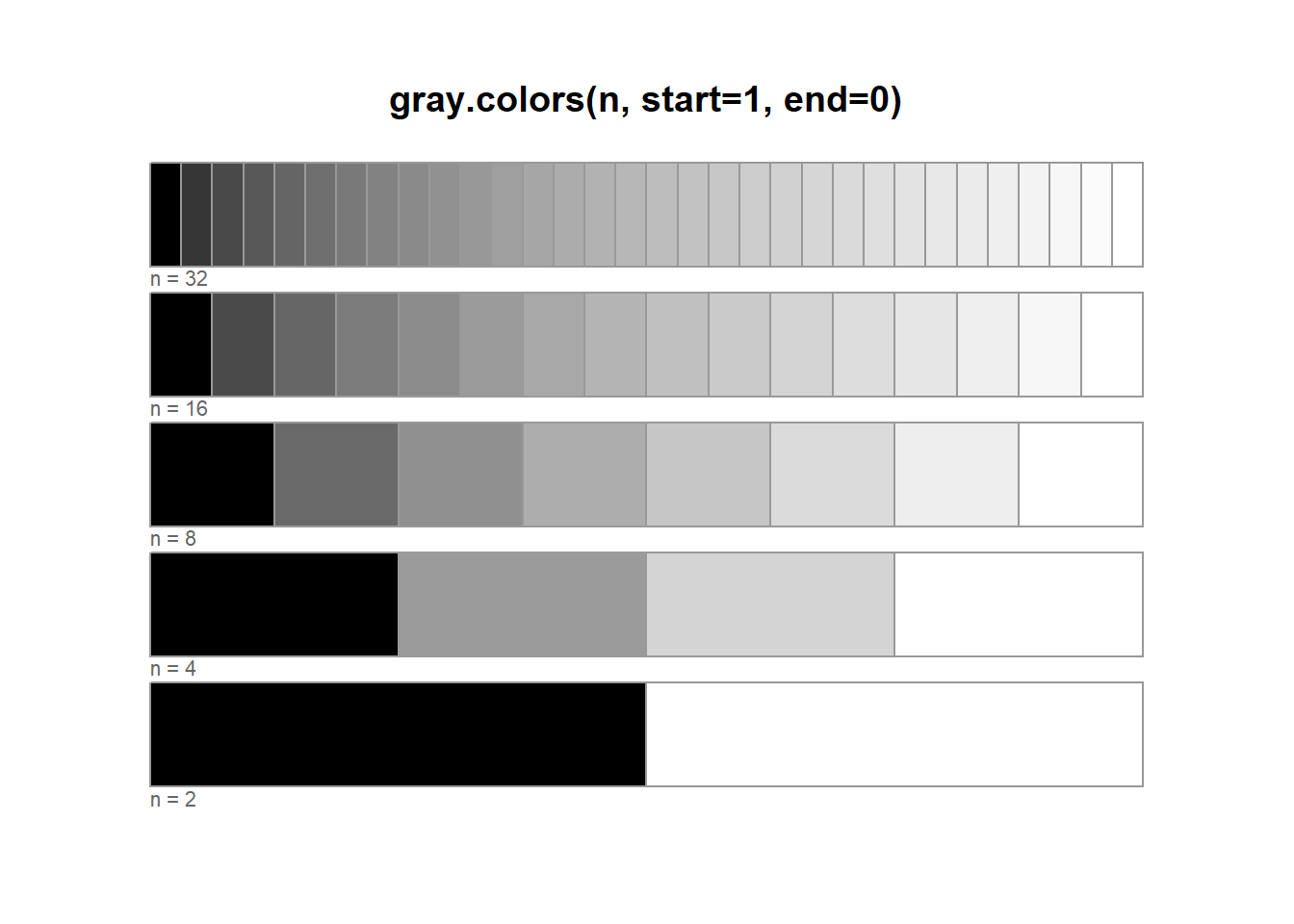
Składnia: gray.colors(num_colors, start=value, end=value, gamma=value).
0 = czerń i 1 = biel (domyślnie start=0.3 i end=0.9).
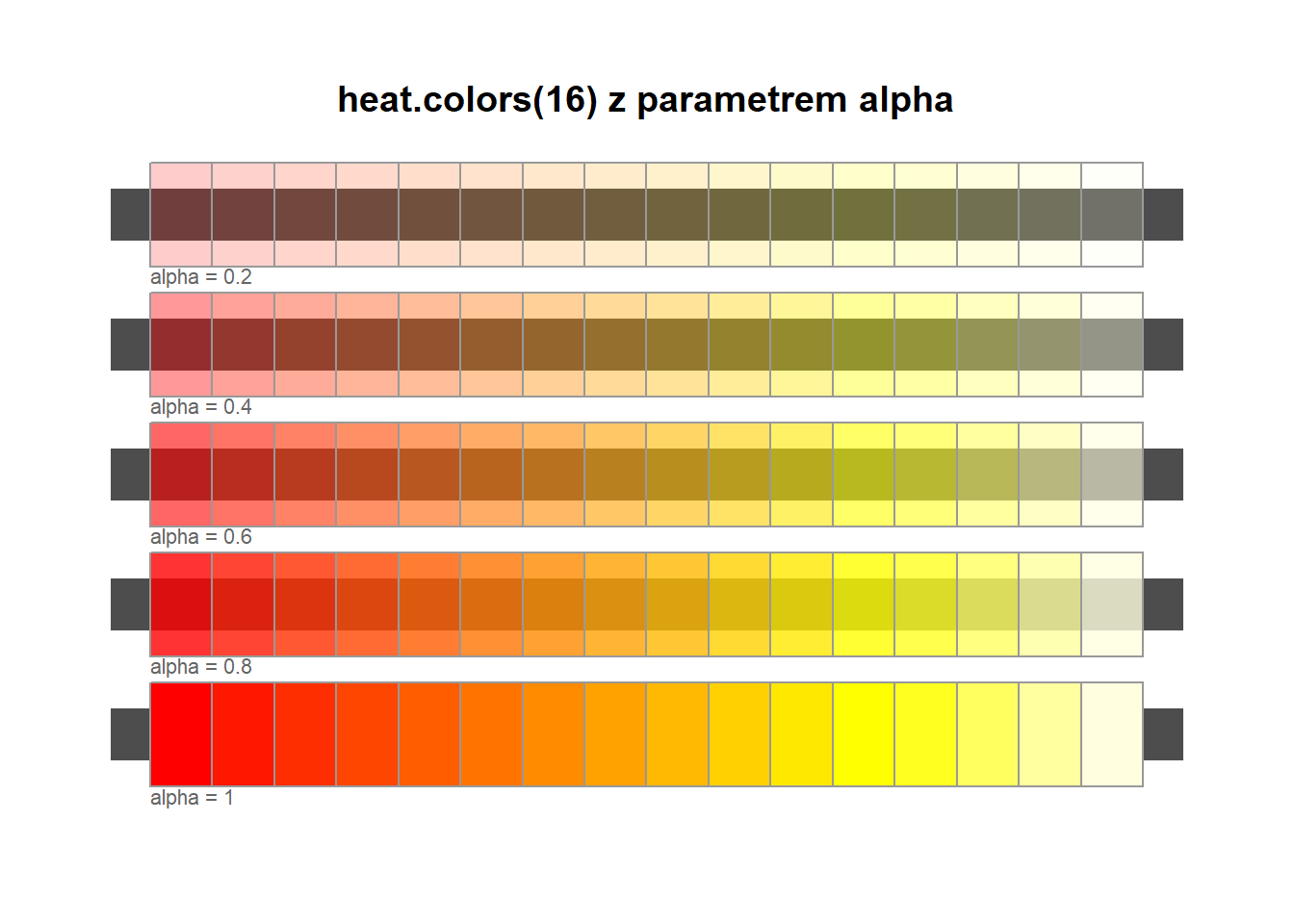
Składnia: heat.colors(num_colors, alpha=value).

Z przezroczystością:
Pozostałe możliwości:

rainbow z określonym początkiem i końcem:
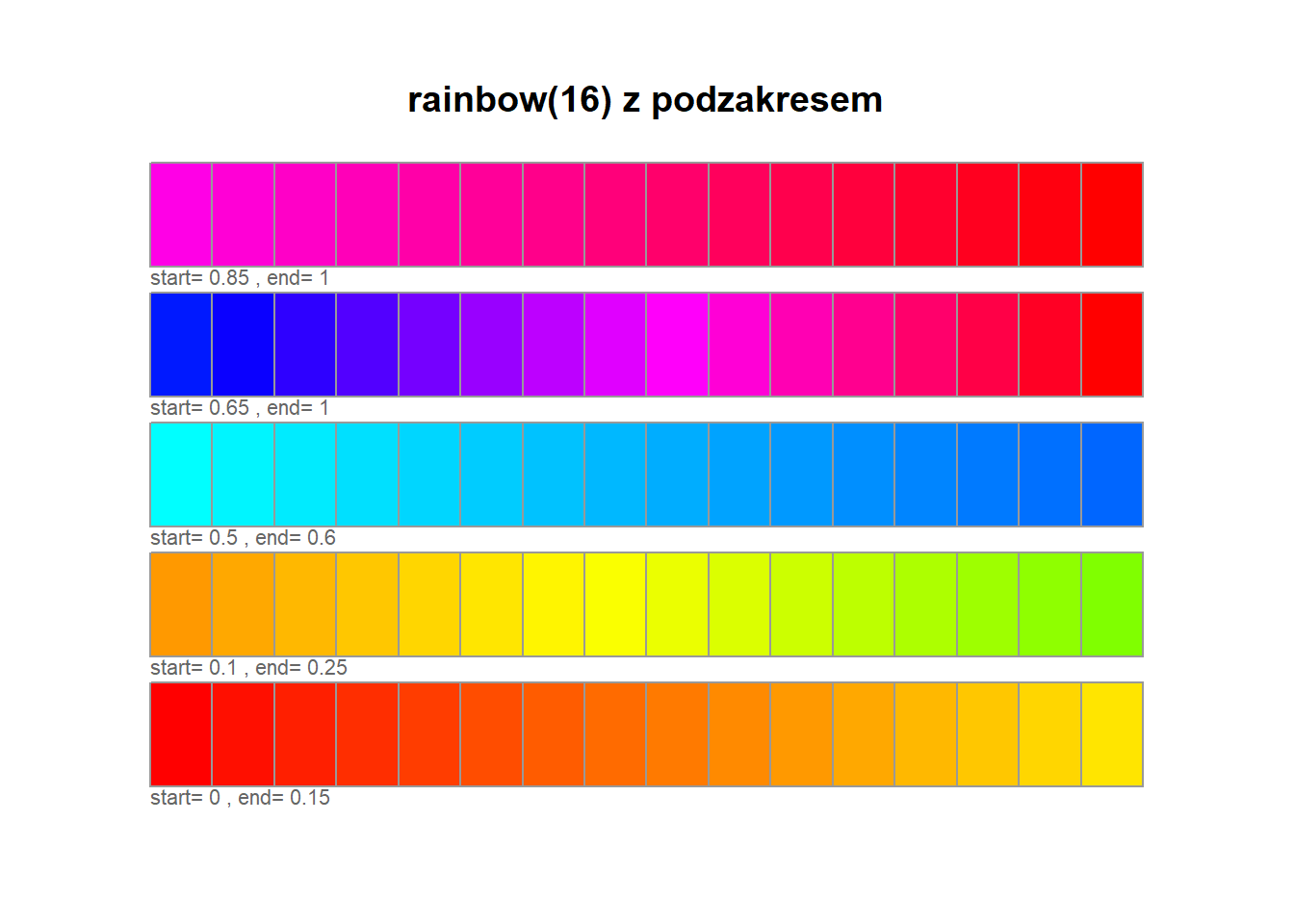
Składnia: hsv(h=value, s=value, v=value, gamma=value, alpha=value). Opis na wiki - link.

Wykres słupkowy - barplot
urlopy<- c(23,3,4,15,8)
barplot(urlopy)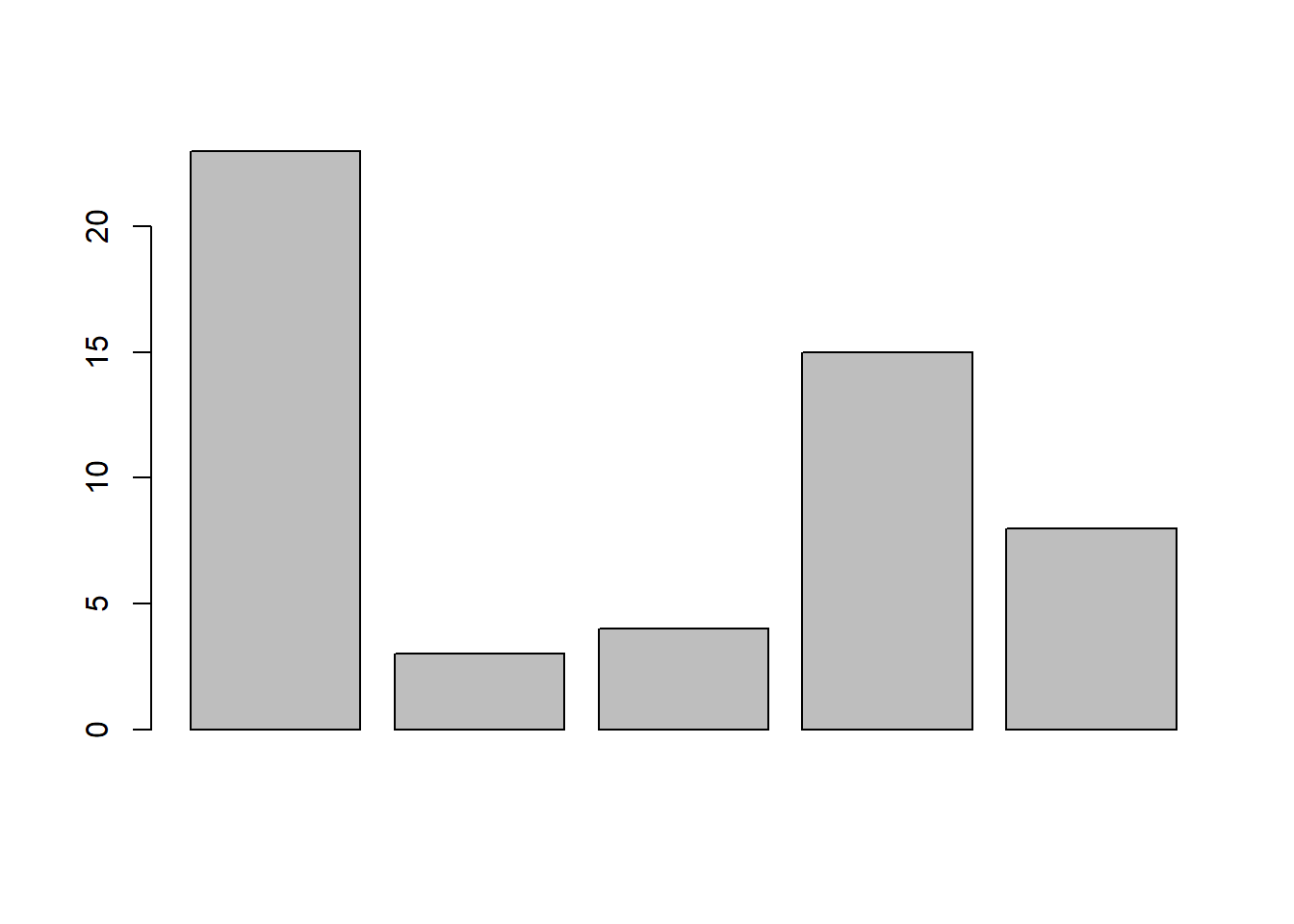
Parametry: names.arg - nazwy argumentów, border - kolor brzegu, density - wypełnienie.
barplot(urlopy, main="Urlopy", xlab="Miesiące",
ylab="Łącznie", names.arg=c("Sty","Lut","Mar","Kwi","Maj"),
border="pink", density=c(10,40,30,20,50))
Parametr horiz=TRUE zmienia orientację na poziomą.
barplot(urlopy, main="Urlopy", xlab="Miesiące",
ylab="Łącznie", names.arg=c("Sty","Lut","Mar","Kwi","Maj"),
border="pink", density=c(10,40,30,20,50), horiz=TRUE)
Parametr beside=TRUE dodaje grupowanie danych (w tym wypadku po kolumnach). bty - typ obramowania (tutaj legendy, n-brak, o- dookoła).
urlopy<- matrix( c(2, 4, 8, 1,4, 5, 7,11,3,24,12,9), nrow=4, ncol=3)
urlopy## [,1] [,2] [,3]
## [1,] 2 4 3
## [2,] 4 5 24
## [3,] 8 7 12
## [4,] 1 11 9barplot(urlopy, main="Urlopy", ylab= "Razem",
beside=TRUE, col=rainbow(4))
legend("topleft", c("1Q","2Q","3Q","4Q"), cex=0.6,
bty="n", fill=rainbow(4))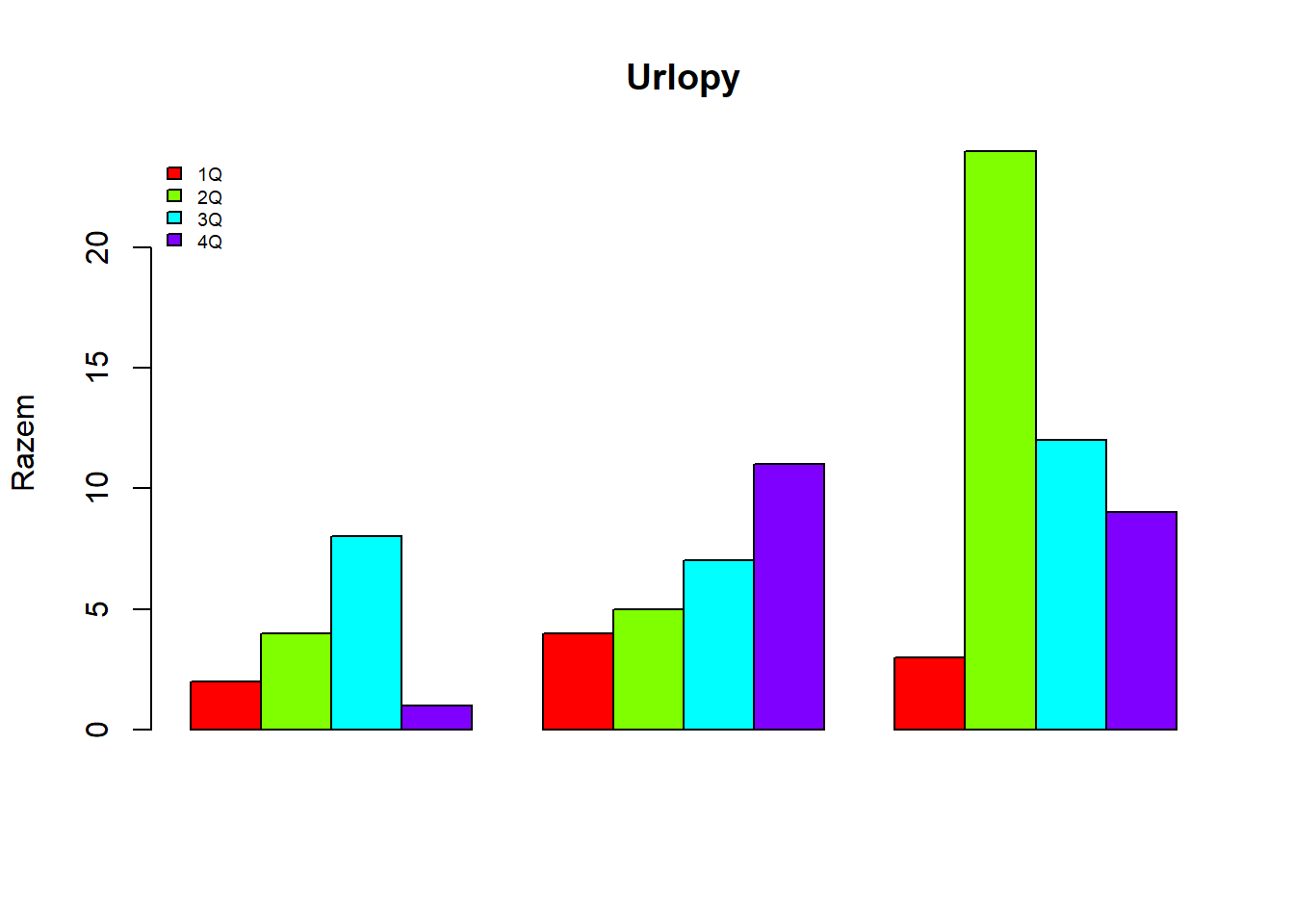
Wykres słupkowy na podstawie macierzy. space - odstęp między słupkami.
barplot(urlopy, main="Urlopy", ylab="Razem",
col=heat.colors(4), space=0.1, cex.axis=0.8, las=1,
names.arg=c("A","B","C"), cex=0.8)
legend("top", c("1Q","2Q","3Q","4Q"), cex=0.8, fill=heat.colors(4));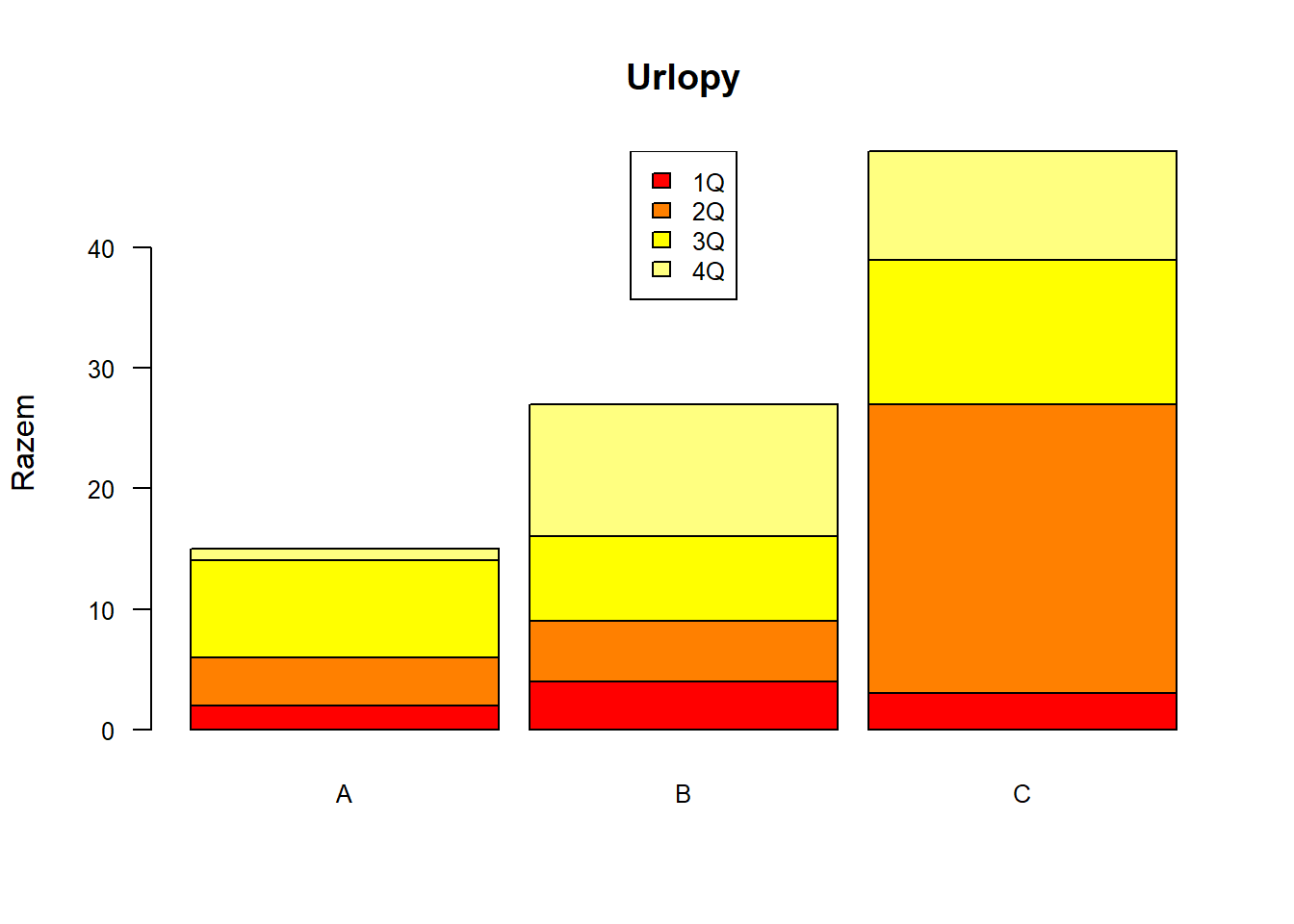
Legenda - jeszcze raz
x <- seq(-2*pi,2*pi,0.1)
plot(x, sin(x),ylab="",type="l",col="blue")
lines(x,cos(x), col="red")
legend("topleft",c("sin(x)","cos(x)"),fill=c("blue","red"))
Mozna też określić współrzędne. Ale musimy być ostrożni, aby nie wypaść poza obszar wykresu.
x <- seq(-2*pi,2*pi,0.1)
plot(x, sin(x),ylab="",type="l",col="blue")
lines(x,cos(x), col="red")
legend(0.5,0.2,c("sin(x)","cos(x)"),fill=c("blue","red"))
Inne przykłady:
x<-1:10; y1=x*x; y2=2*y1
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
lines(x, y2, pch=18, col="blue", type="b", lty=2)
legend(1, 95, legend=c("Line 1", "Line 2"),
col=c("red", "blue"), lty=1:2, cex=0.8)
x<-1:10; y1=x*x; y2=2*y1
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
lines(x, y2, pch=18, col="blue", type="b", lty=2)
legend(1, 95, legend=c("Line 1", "Line 2"),
col=c("red", "blue"), lty=1:2, cex=0.8,
title="Line types", text.font=4, bg='lightblue')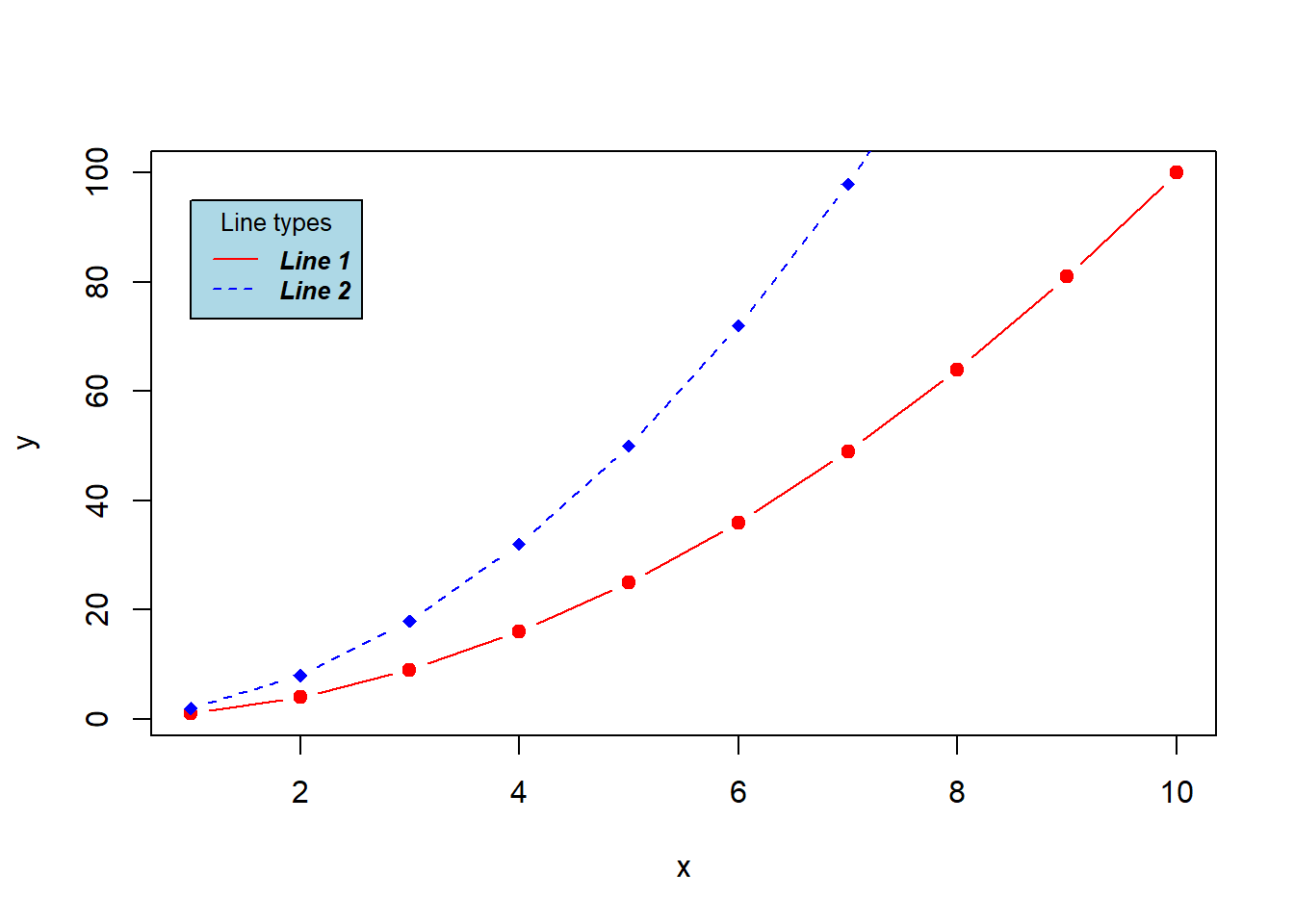
x<-1:10; y1=x*x; y2=2*y1
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
lines(x, y2, pch=18, col="blue", type="b", lty=2)
legend(1, 95, legend=c("Line 1", "Line 2"),
col=c("red", "blue"), lty=1:2, cex=0.8,
box.lty=2, box.lwd=2, box.col="green")
x<-1:10; y1=x*x; y2=2*y1
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
lines(x, y2, pch=18, col="blue", type="b", lty=2)
legend(1, 95, legend=c("Line 1", "Line 2"),
col=c("red", "blue"), lty=1:2, cex=0.8,
box.lty=0)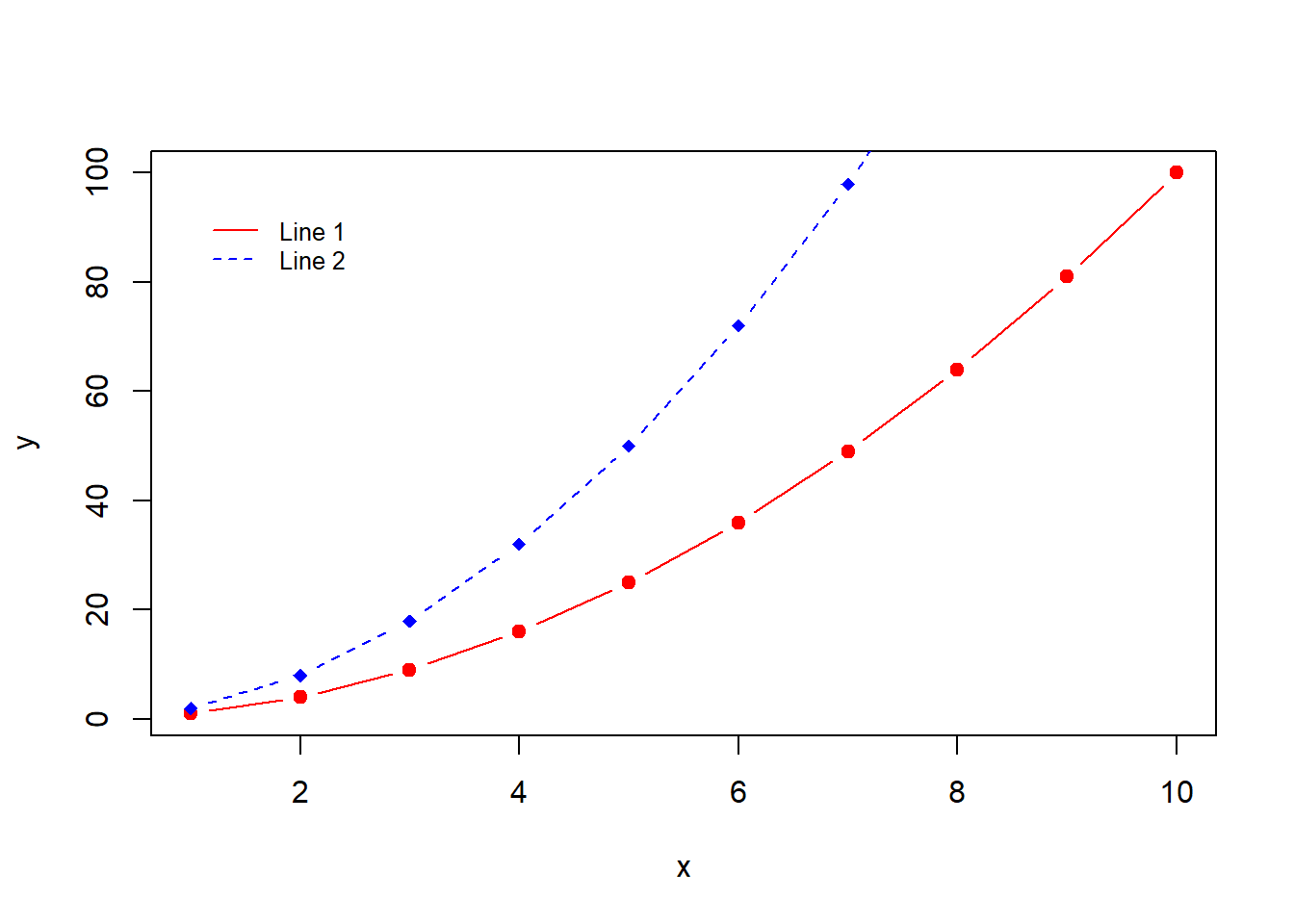
boxplot(mtcars$mpg~mtcars$cyl,
xlab="Cylinders", ylab="Miles/(US) gallon",
col=topo.colors(3))
legend("bottomleft", inset=.02, title="Number of Cylinders",
c("4","6","8"), fill=topo.colors(3), horiz=TRUE, cex=0.8)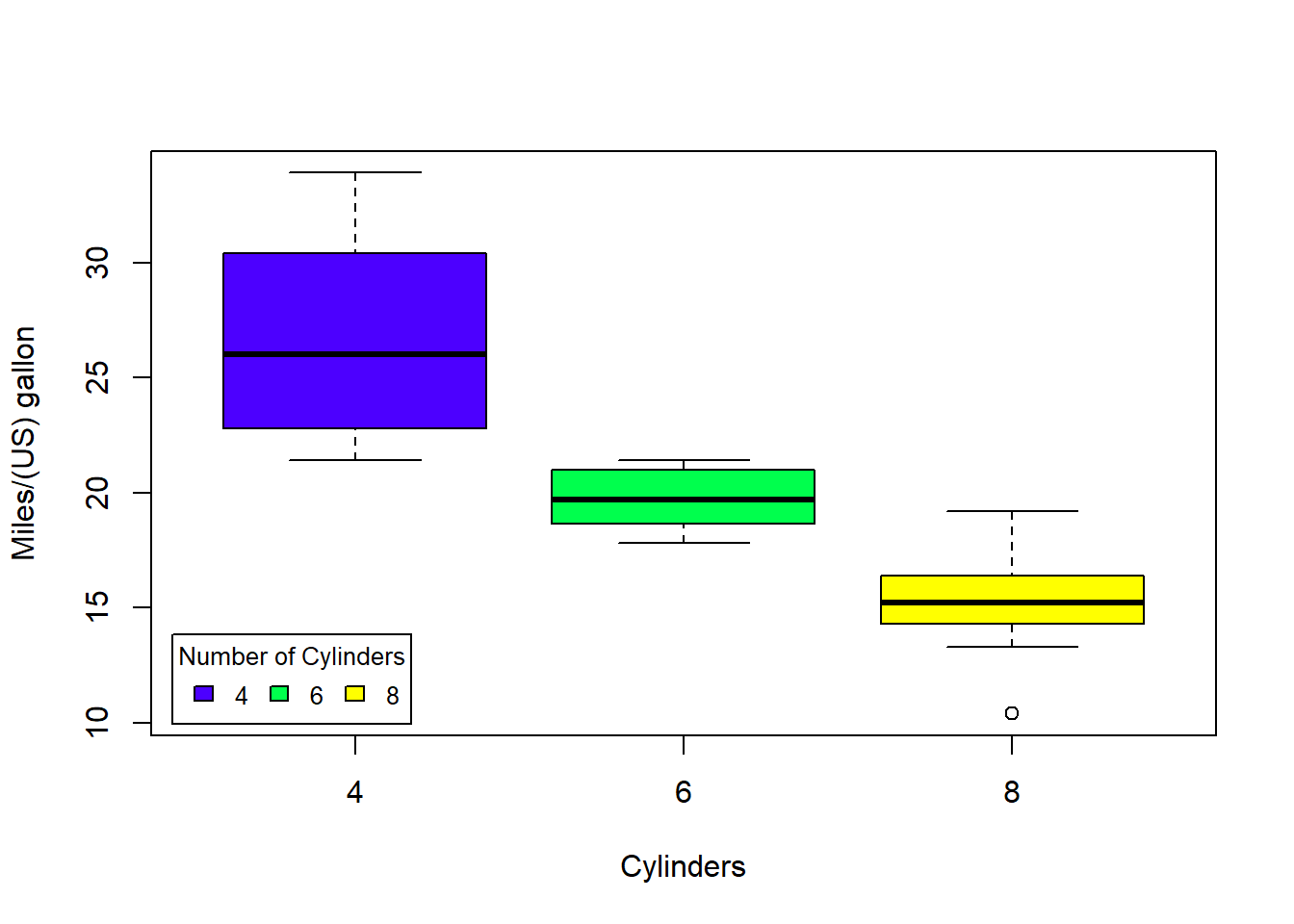
Generowanie rozkładu normalnego
Składnia: rnorm(n, mean = 0, sd = 1). Jako wynik otrzymujemy wektor n obserwacji.
Histogram i wykres gęstości
Parametr prob = TRUE odpowiada za wyświetlanie gęstości a nie liczebności.
x<-rnorm(1000)
hist(x, prob = TRUE)
lines(density(x), xlab="", ylab="", main="")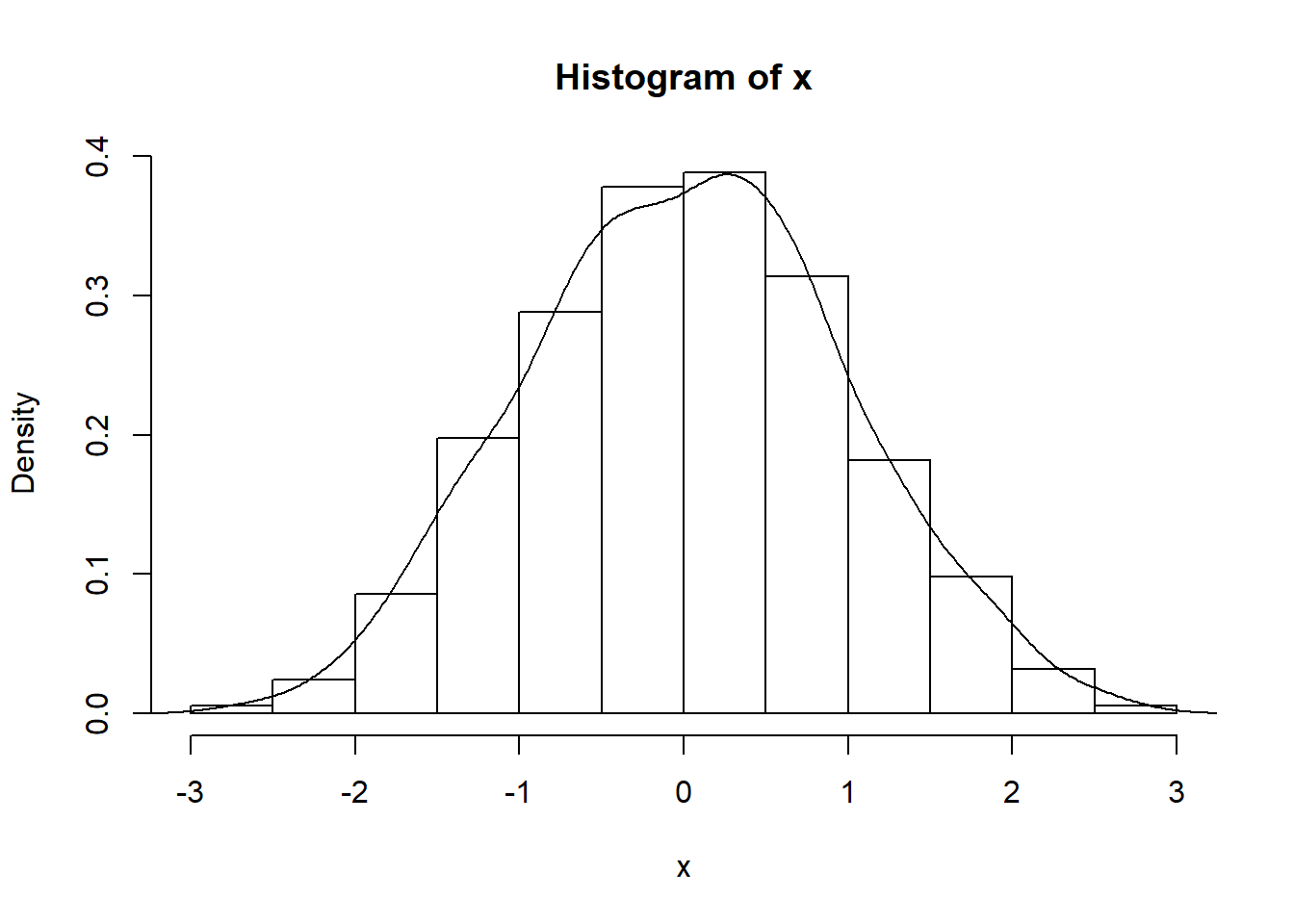
breaks określa punkty podziałów. Może być liczbą (ale zaokrąglenie do “piątek”).
hist(AirPassengers, main="Pasażerowie linii lotniczych", xlab="Pasażerowie",
border="blue", col="red", xlim=c(100,700), las=1,
breaks=9)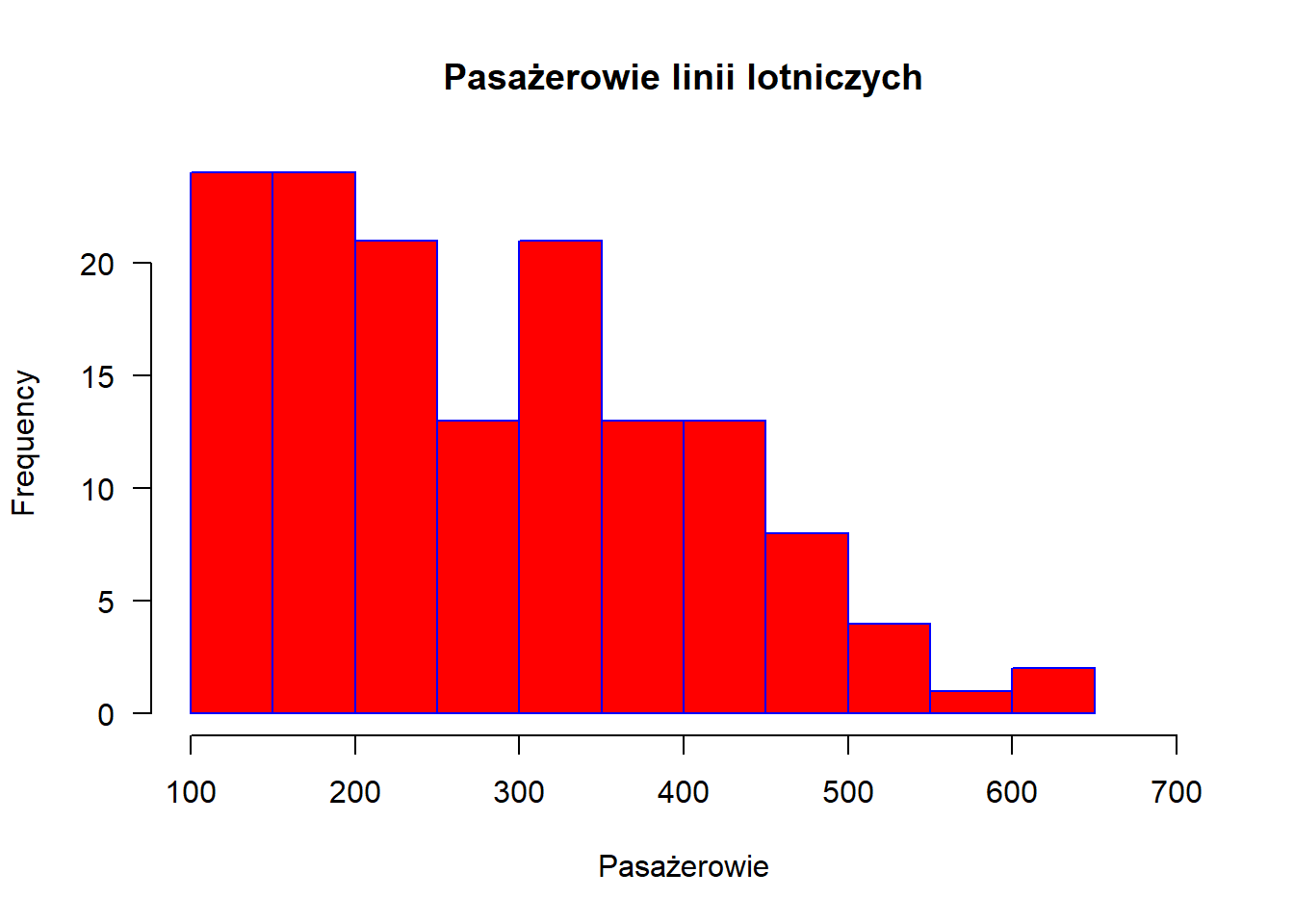
Druga opcja to podanie wektora. Ale ostrożnie.
hist(AirPassengers, main="Pasażerowie linii lotniczych", xlab="Pasażerowie",
border="blue", col="red", xlim=c(100,700), las=1,
breaks=c(100,300,400,500,650))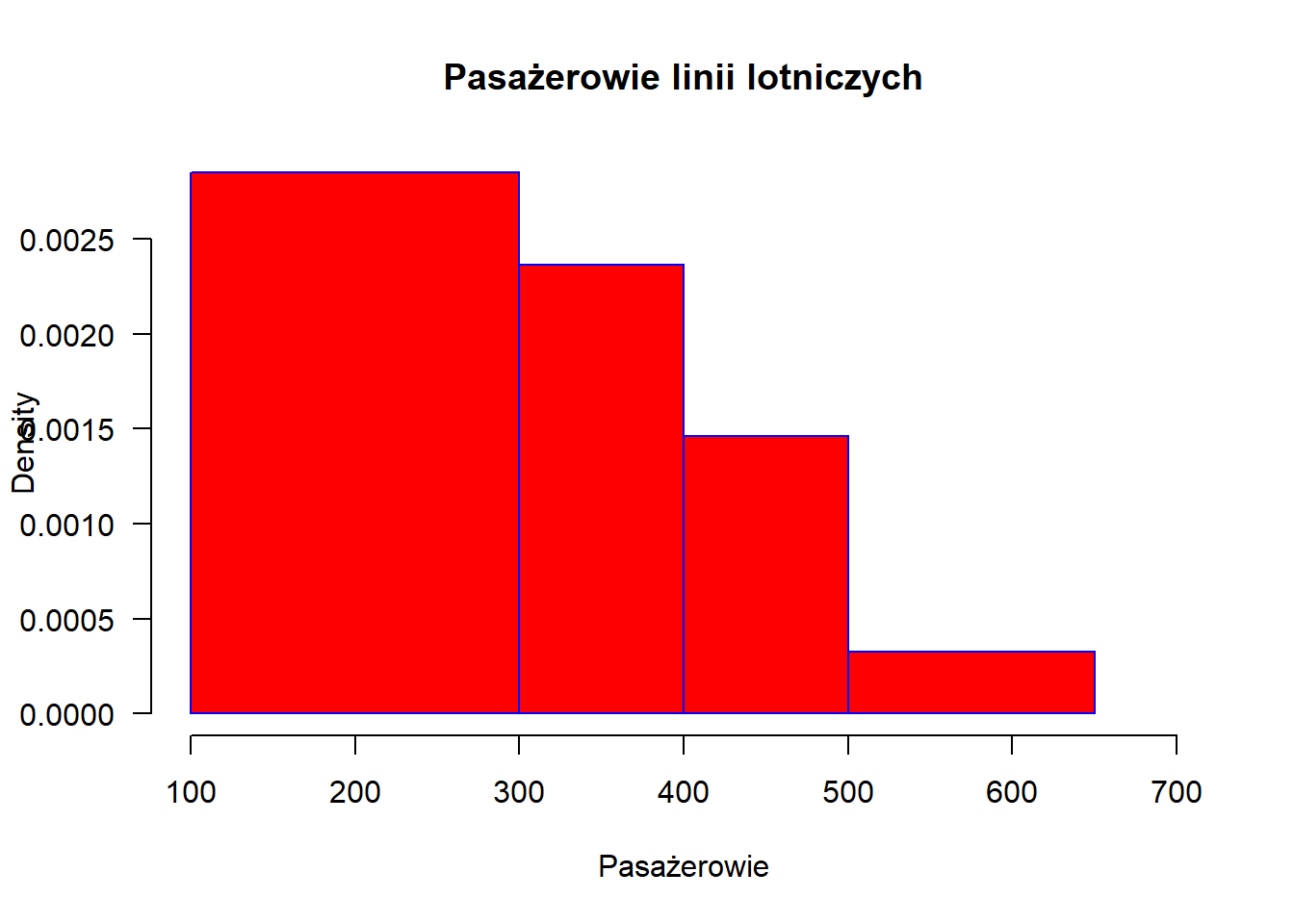
hist(AirPassengers, main="Pasażerowie linii lotniczych", xlab="Pasażerowie",
border="blue", col="red", xlim=c(100,700), las=1,
breaks=c(100,300,500,700))
Wykres pudełkowy - boxplot
x<-c(3,4,5,6,7,8,1,2,3,14)
boxplot(x)
Przy dwóch zmiennych możemy użyć poniższej składni:
boxplot(mpg~cyl,data=mtcars, main="Dane o samochodach",
xlab="Liczba cylindrów", ylab="Mile na galon")
Parametr horizontal=TRUE zmienia orientację na poziomą. notch- dodaje “zwężenie”.
x<-c(3,4,5,6,7,3,5,6,7,3,1,2,10,3,20)
boxplot(x, horizontal = TRUE, notch=TRUE)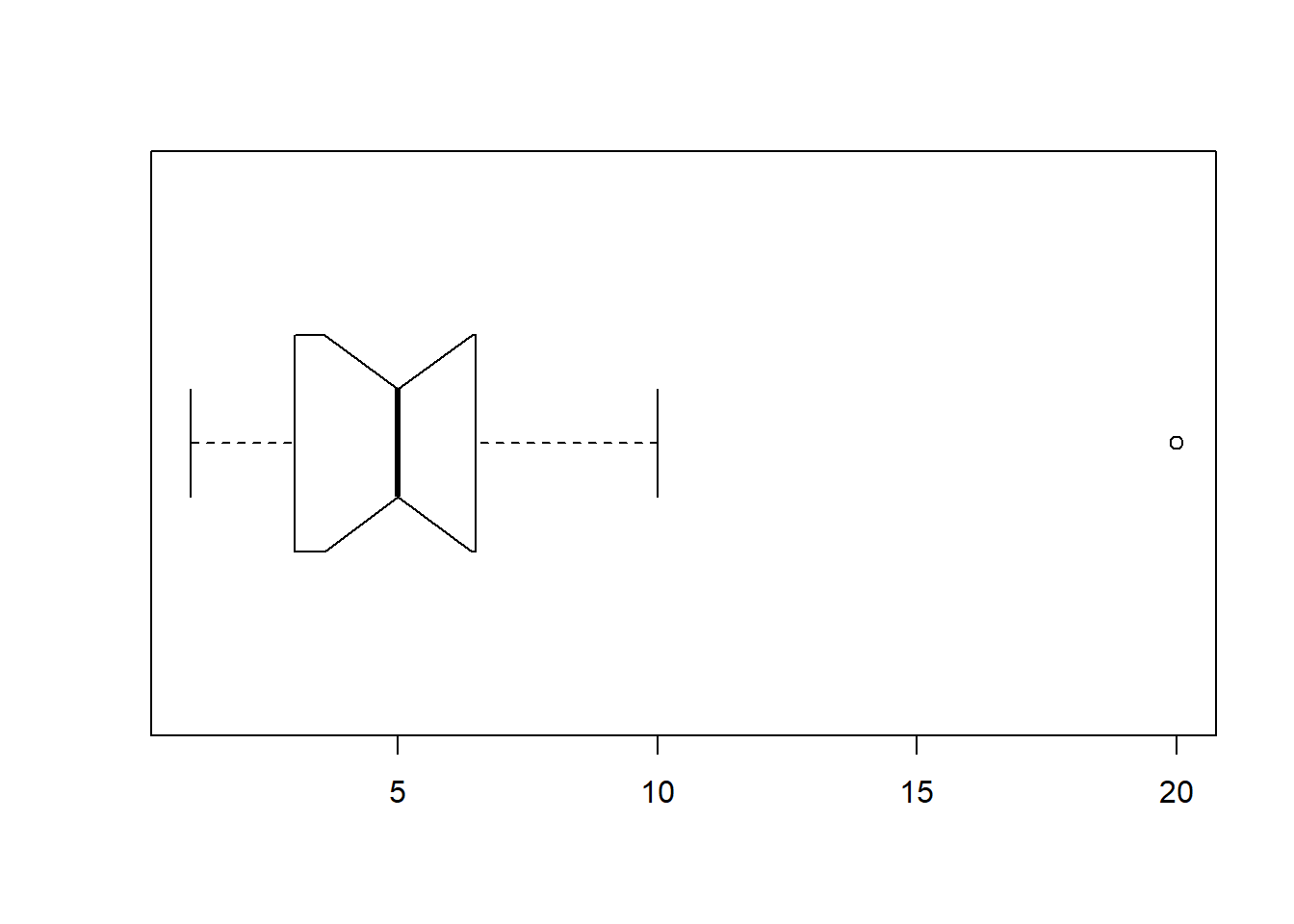
Mapy ciepła
data=as.matrix(mtcars)
heatmap(data)
heatmap(data, scale="column")
heatmap(data, Colv = NA, Rowv = NA, scale="column")
Wykres punktowy dla trzech zmiennych
Uwaga: warto zwrócić uwagę na parametry kolorów.
library(MASS)
head(ships)## type year period service incidents
## 1 A 60 60 127 0
## 2 A 60 75 63 0
## 3 A 65 60 1095 3
## 4 A 65 75 1095 4
## 5 A 70 60 1512 6
## 6 A 70 75 3353 18plot(ships$incidents,ships$service,col=ships$type, pch=16)
legend("topleft",legend = levels(ships$type), col = c(1:5), pch=16)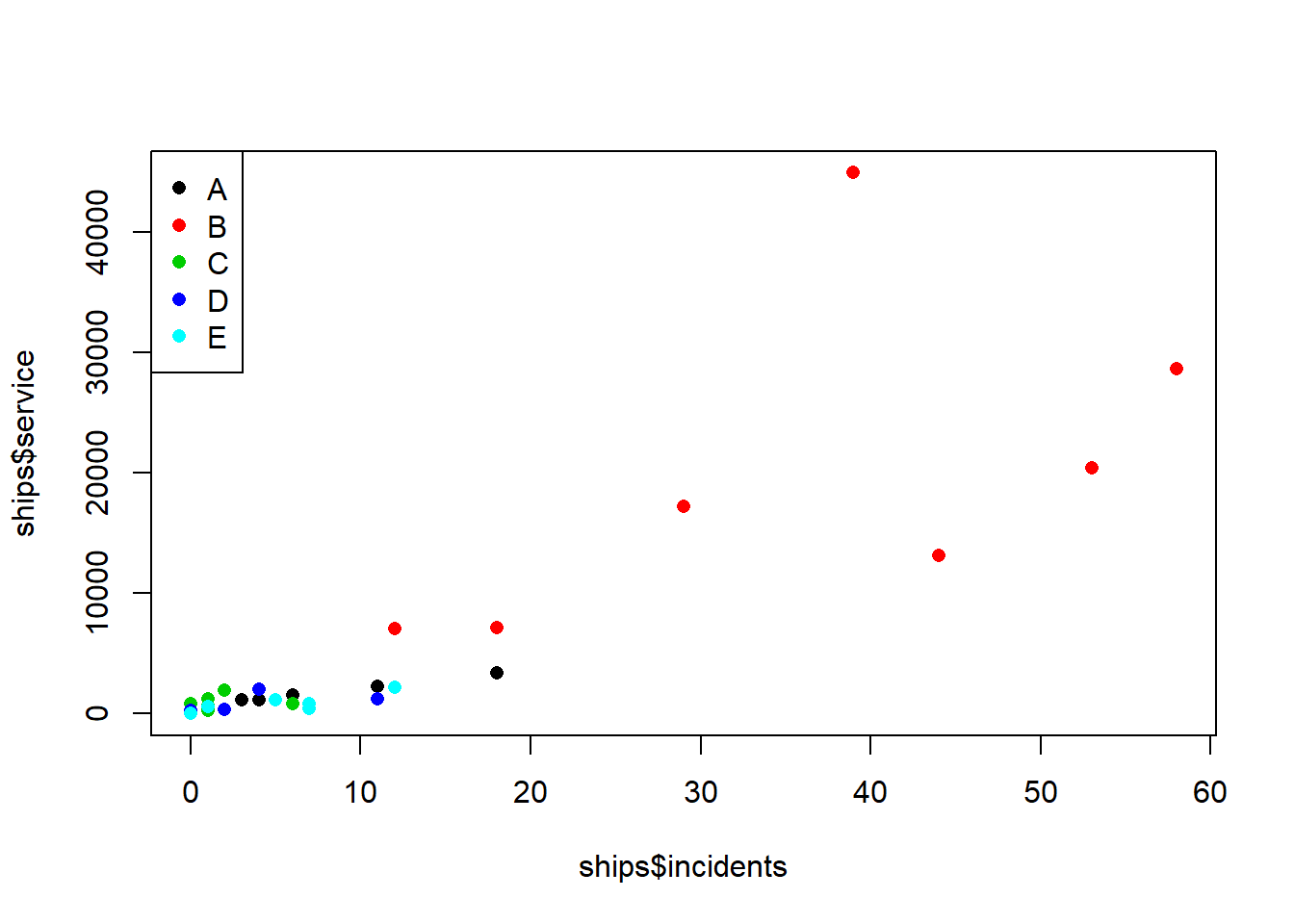
Baza iris
data(iris)
head(iris)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosaplot(iris$Sepal.Length, iris$Petal.Length, col = iris$Species)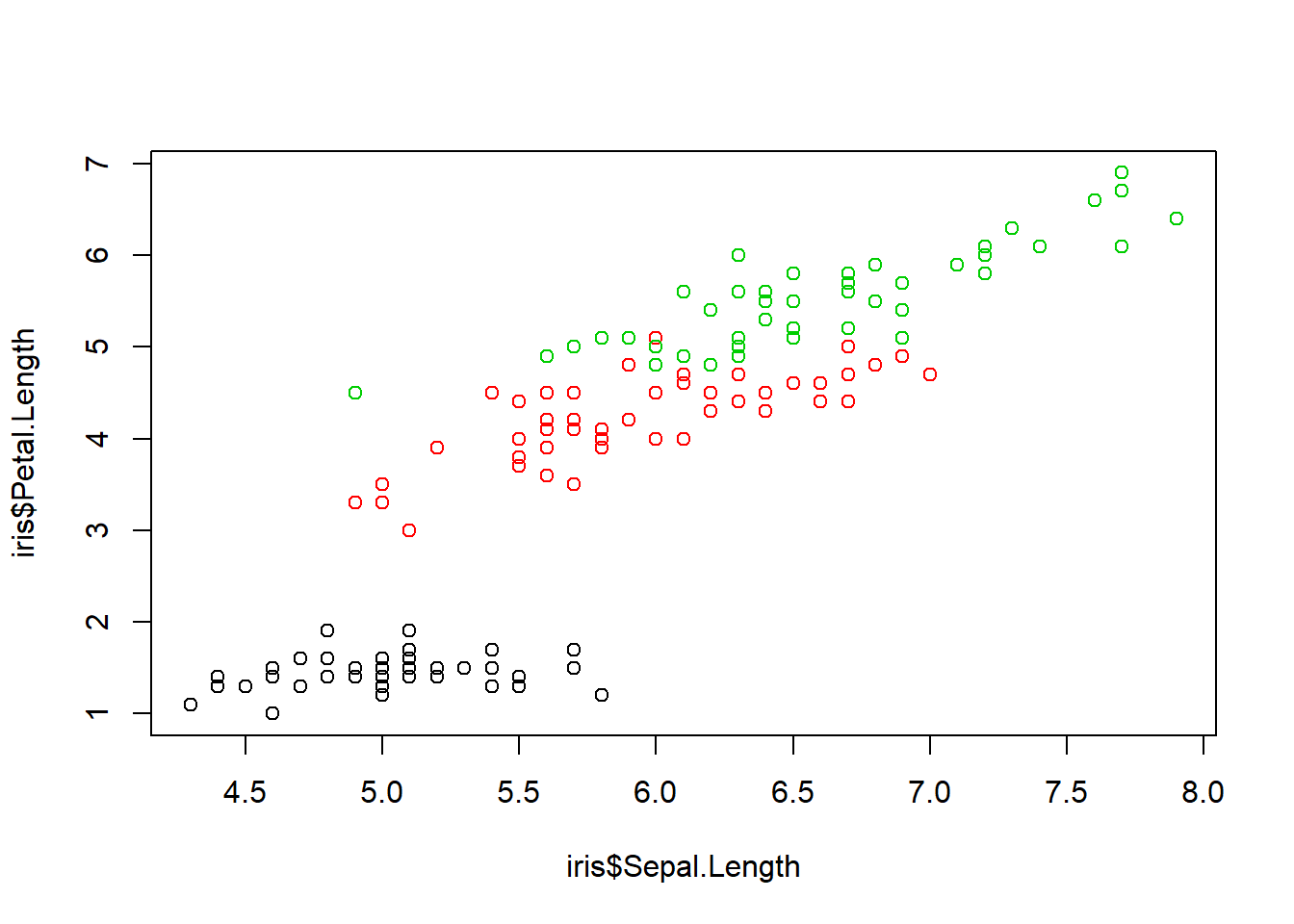
plot(iris$Sepal.Length, iris$Petal.Length, col = iris$Species, pch = 16)
plot(iris$Sepal.Length, iris$Petal.Length,
col = iris$Species,
pch = 16,
cex = 2)
legend(x = 4.5, y = 7, legend = levels(iris$Species), col = c(1:3), pch = 16)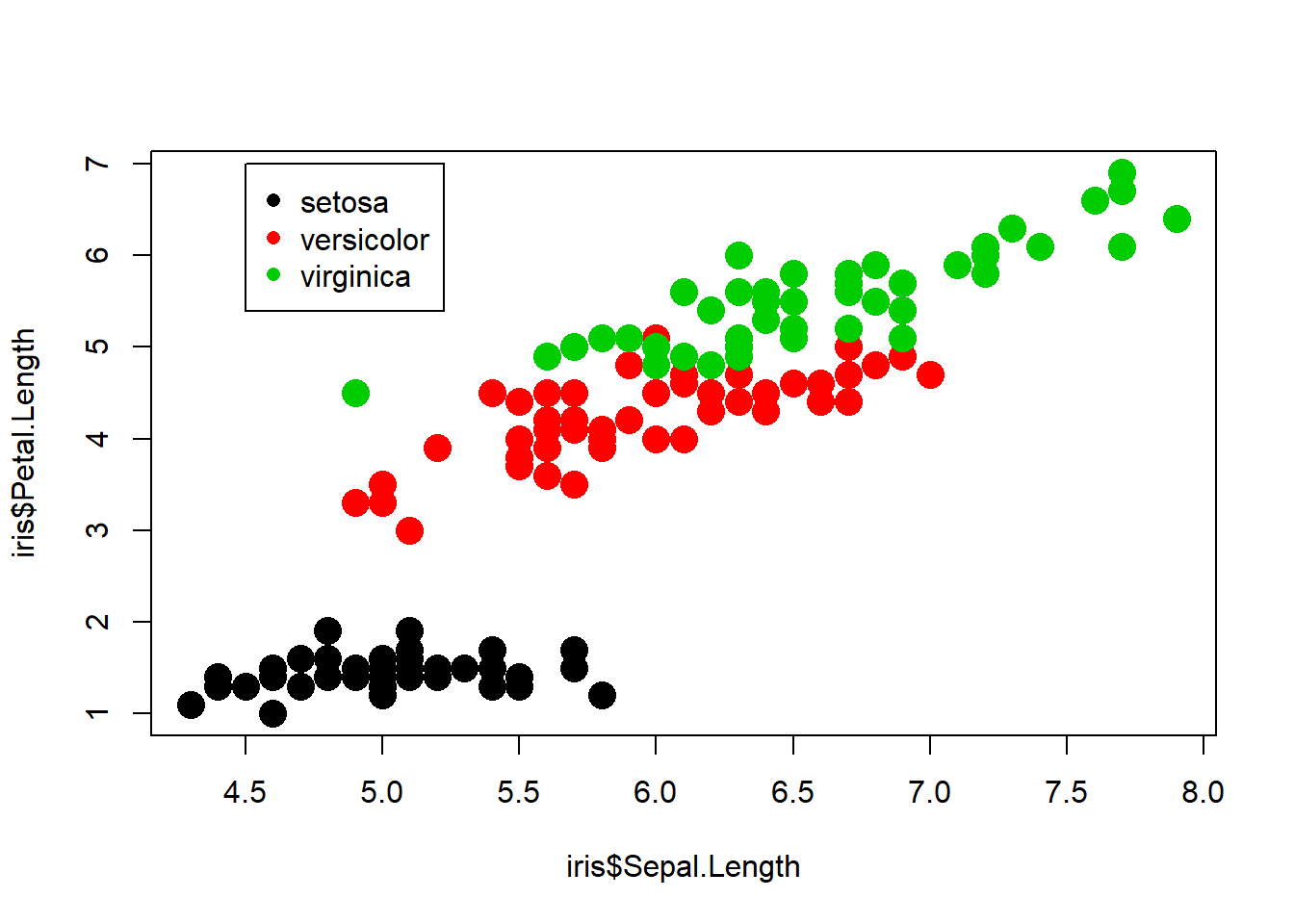
pairs(iris)
Wykres mozaikowy
W tym typie wykresu podtrzebujemy zwykle macierzy/tabeli.
choroby<-matrix(c(34,12,22,18),ncol=2,byrow=TRUE)
colnames(choroby) <- c("M","K")
rownames(choroby)<- c("GDO", "DDO")
mosaicplot(choroby, color = rainbow(2))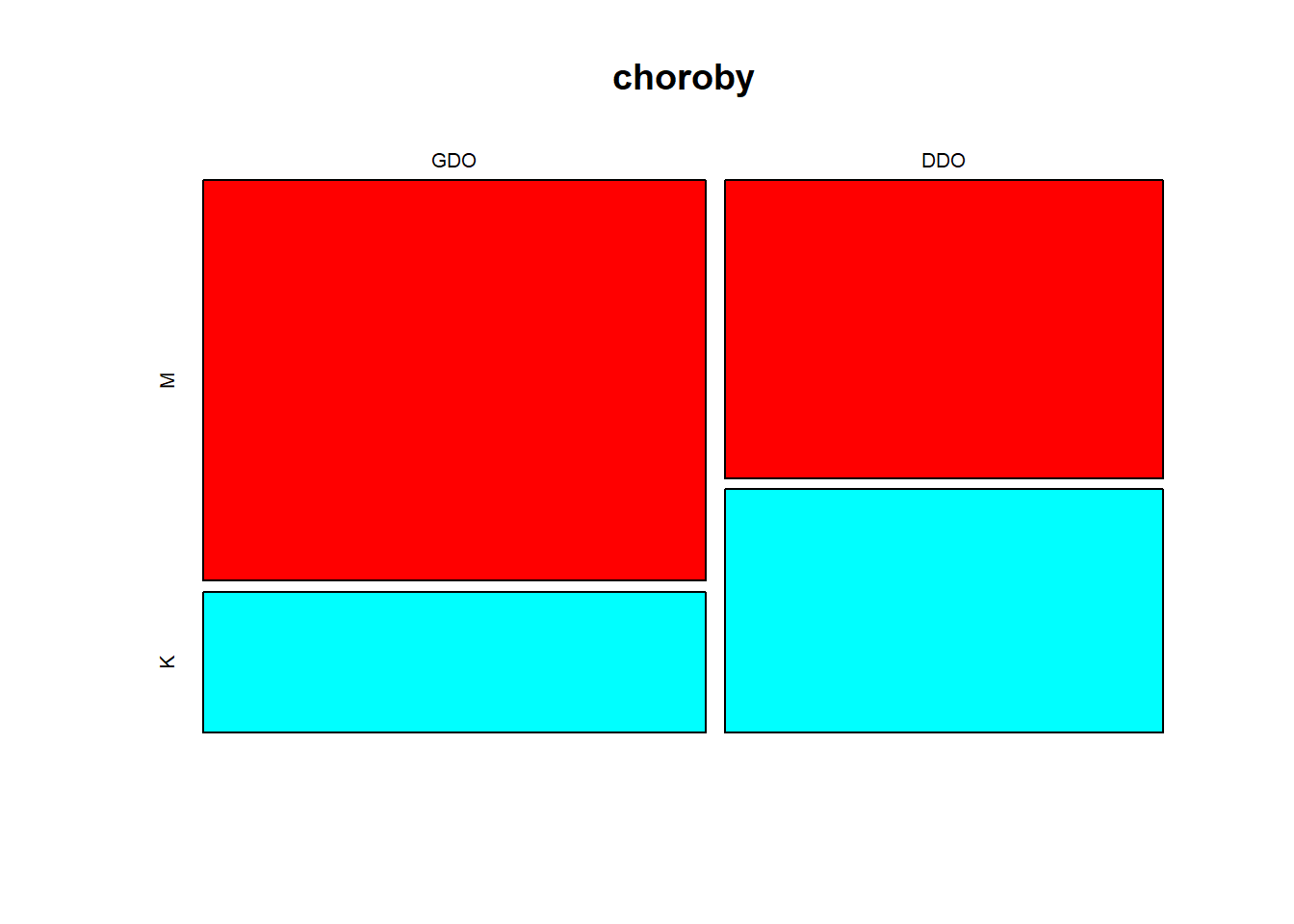
mosaicplot(HairEyeColor, col=heat.colors(2))
Wykres w perspektywie
x <- seq(-10, 10, length = 30)
y <- x
f <- function(x, y) {r <- sqrt(x ^ 2 + y ^ 2); 10 * sin(r) / r}
z <- outer(x, y, f)
persp(x, y, z)
Mapy - cd.
library(raster)## Loading required package: sp##
## Attaching package: 'raster'## The following objects are masked from 'package:MASS':
##
## area, selectlibrary(sp)
map1<- getData('GADM', country='POL', level=2)
map2 <- map1[map1$NAME_1=="Warmińsko-Mazurskie",]
plot(map2)
Strzałki i wykresy z błędami
Arrows - link do dokumentacji - link.
plot(mpg~disp,data=mtcars)
arrows(x0=mtcars$disp,
y0=mtcars$mpg*0.95,
x1=mtcars$disp,
y1=mtcars$mpg*1.05,
angle=90,
code=3,
length=0.04,
lwd=0.4)
a<-c(3,4.5,5)
b<-c(0.1,0.2,0.3)
x<-data.frame(a,b)
colnames(x)<-c("pomiar","blad")
plot(x$pomiar, pch=19, ylim=c(2.5,5.5))
arrows( 1:dim(x)[1],x$pomiar-x$blad,1:dim(x)[1], x$pomiar+x$blad,
angle=90, code=3,
length=0.04)